Membres de l'équipe
Mécanismes de régulation des gènes par les facteurs de transcription
Nous explorons les mécanismes de régulation de la transcription qui régissent la régulation du génome à l'interface des facteurs de transcription, des protéines de la chromatine et de la machinerie de transcription, en encadrant les résultats dans les processus de développement.
La régulation des gènes est au cœur du destin cellulaire et dépend fortement de l’activité des facteurs de transcription. On sait peu de choses sur la façon dont les complexes de régulation de la transcription s’assemblent, comment ils négocient la chromatine et ce qui sous-tend leur spécificité fonctionnelle et leur diversité. Nos projets visent à explorer les mécanismes de régulation de la transcription pour, à terme, mieux comprendre la régulation du génome. Nous nous concentrons sur les facteurs de transcription Hox, une famille de facteurs de transcription à homéodomaine ayant des fonctions clés dans le développement, l’évolution et les processus physio-pathologiques.
Les travaux de l’équipe ont contribué à clarifier le paradoxe de la spécificité de Hox, en identifiant les séquences protéiques intrinsèques de Hox responsables de la spécificité, et en découvrant de nouveaux modes d’interactions avec les cofacteurs de spécificité de la classe PBC. Nous avons également identifié des liens physiques et fonctionnels avec les protéines de la chromatine, notamment les protéines du complexe PcG et Mediator, ainsi que des liens avec le facteur de pause de la transcription M1BP, reliant l’activité des protéines Hox à la modification de la chromatine et à l’activité de la machinerie basale de transcription.
Plus récemment, nous avons découvert une nouvelle facette de la fonction des protéines Hox, où les protéines Hox agissent de manière non spécifique aux paralogues, une propriété qui correspond mieux aux propriétés biochimiques Hox partagées. Nos directions de recherche actuelles visent à étudier au niveau physiologique comment les fonctions spécifiques et non spécifiques de Hox sont liées les unes aux autres, en explorant les fonctions des protéines Hox dans deux tissus, le corps gras larvaire et le développement musculaire adulte. Les travaux visent également à découvrir les principes moléculaires des fonctions génériques de Hox, qui n’ont jusqu’à présent pas été étudiés.
Publications
M1BP is an essential transcriptional activator of oxidative metabolism during Drosophila development
Hox Proteins in the Regulation of Muscle Development
Hox functional diversity: Novel insights from flexible motif folding and plastic protein interaction
Hox proteins mediate developmental and environmental control of autophagy
M1BP is an essential transcriptional activator of oxidative metabolism during Drosophila development
Hox Proteins in the Regulation of Muscle Development
Fattening the perspective of Hox protein specificity through SLiMming
Post-translational modifications of HOX proteins, an underestimated issue
Hox functional diversity: Novel insights from flexible motif folding and plastic protein interaction
Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition).
Hox genes : a fertile interplay of concepts and methods.
Hox proteins mediate developmental and environmental control of autophagy
Distinct genetic requirements for BX-C mediated specification of abdominal denticles.
Reiterative use of signalling pathways controls multiple cellular events during Drosophila posterior spiracle organogenesis.
Reptin and Pontin function antagonistically with PcG and TrxG complexes to mediate Hox gene control.
Ectopic Myf5 or MyoD prevents the neuronal differentiation program in addition to inducing skeletal muscle differentiation, in the chick neural tube
Purification and characterization of mSin3A-containing Brg1 and hBrm chromatin remodeling complexes.
Delta 1-activated notch inhibits muscle differentiation without affecting Myf5 and Pax3 expression in chick limb myogenesis
pannier acts upstream of wingless to direct dorsal eye disc development in Drosophila
Wnt and TGFbeta signals subdivide the AbdA Hox domain during Drosophila mesoderm patterning.
RING1 is associated with the polycomb group protein complex and acts as a transcriptional repressor.
Actualités
Plusieurs projets portés par nos équipes ont été sélectionnés pour un financement par l’ANR et la FRM, mettant en lumière leur travail et leur recherche innovante.
Réalisations et promotions récentes
Fais ton stage à l’IBDM !
Tu es à la recherche de ton stage de Master ? L’IBDM te semble être le bon endroit pour le faire ? Découvre nos offres dès maintenant.
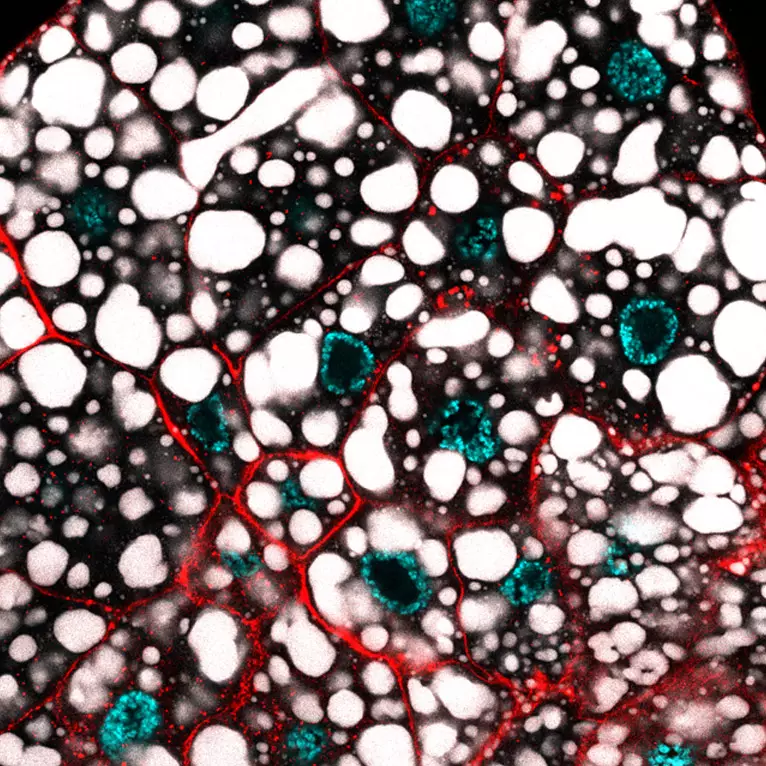
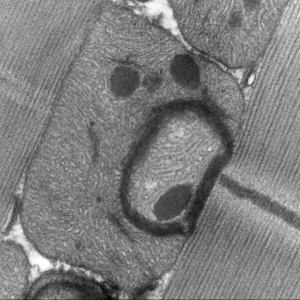
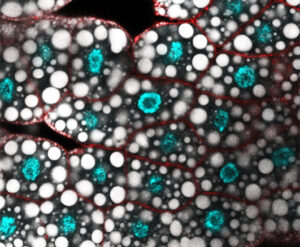
En utilisant le muscle de vol de la drosophile, le groupe de Saurin et Graba identifie le facteur de transcription M1BP comme un nouveau régulateur majeur du métabolisme oxydatif mitochondrial.
Identification d’une fonction générique des protéines Hox dans le tube neural chez les vertébrés
L’équipe de Yacine Graba et Andrew Saurin vient d’identifier une fonction atypique des gènes HoxB au cours du développement de la moelle épinière.
How two recently discovered and oppositely acting transcriptional regulators control metabolism in the Drosophila larval fat body, with a special attention on metabolic paths linked to fat accumulation.
Les travaux porteront sur la manière dont les séquences de protéines de facteurs de transcription influencent la séparation de phase liquide-liquide (LLPS), la sous-compartimentation nucléaire et la régulation des gènes au cours du développement.
The project aims at uncovering the molecular and cellular bases for Ubx and AbdA distinct motif usage.
The M2 project will use state-of-the-art genomics profiling techniques, Drosophila genetics, imaging and molecular biology for studying the antagonistic transcriptional control and dysfunction of fat body lipid metabolism development.
The M2 project will use state-of-the-art genomics profiling techniques, Drosophila genetics, imaging and molecular biology for studying muscle development.
Membres de l'équipe
Alumni
Les organismes qui nous financent